| 항목 | 고정효과 선형모델 (고정효과) ANOVA | 고정효과 선형모델 (고정효과) BLUE | 랜덤효과 선형모델 (랜덤효과) BLUP | 혈통 혼합선형모델 (고정효과+랜덤효과) PBLUP | 유전체 혼합선형모델 (고정효과+랜덤효과) GBLUP | 유전 혼합선형모델 (고정효과+랜덤효과) ssGBLUP | 설명 |
|---|---|---|---|---|---|---|---|
| 스칼라 모델식 | $y_{ij} = \mu + \alpha_i + \varepsilon_{ij}$ $(i=1,\ldots,k;\ j=1,\ldots,n_i)$ | $y_i =\mathbf{x}_i^{\top}\boldsymbol{\beta} + \varepsilon_i$ $(i=1,\ldots,n)$ | $y_i = \mu + \mathbf{z}_i^{\top}\mathbf{u} + \boldsymbol{\varepsilon}_i$ $(i=1,\ldots,n)$ | $y_i = \mathbf{x}_i^{\top}\boldsymbol{\beta} + \mathbf{z}_{a,i}^{\top}\mathbf{a} + \varepsilon_i$ $\qquad (i=1,\ldots,n)$ | $y_i = \mathbf{x}_i^{\top}\boldsymbol{\beta} + \mathbf{z}_{g,i}^{\top}\mathbf{g} + \varepsilon_i$ $(i=1,\ldots,n)$ | $y_i = \mathbf{x}_i^{\top}\boldsymbol{\beta} + \mathbf{z}_i^{\top}\mathbf{u} + \varepsilon_i$ $(i=1,\ldots,n)$ | |
| 벡터 모델식 | $\mathbf{y} = \mathbf{X}\boldsymbol{\beta} + \boldsymbol{\varepsilon}$ | $\mathbf{y} = \mathbf{X}\boldsymbol{\beta} + \boldsymbol{\varepsilon}$ | $\mathbf{y} = \mu \mathbf{1} + \mathbf{Z}\mathbf{u} + \boldsymbol{\varepsilon}$ | $\mathbf{y} = \mathbf{X}\boldsymbol{\beta} + \mathbf{Z_a}\mathbf{a} + \boldsymbol{\varepsilon}$ | $\mathbf{y} = \mathbf{X}\boldsymbol{\beta} + \mathbf{Z_g}\mathbf{g} + \boldsymbol{\varepsilon}$ | $\mathbf{y} = \mathbf{X}\boldsymbol{\beta} + \mathbf{Z}\mathbf{u} + \boldsymbol{\varepsilon}$ | |
| 오차항 분포 | $\boldsymbol{\varepsilon} \sim \mathcal{N}(\mathbf{0}, \sigma^2_{\varepsilon}\mathbf{I})$ | $\boldsymbol{\varepsilon} \sim \mathcal{N}(\mathbf{0}, \sigma^2_{\varepsilon}\mathbf{I})$ | $\boldsymbol{\varepsilon} \sim \mathcal{N}(\mathbf{0}, \sigma^2_{\varepsilon}\mathbf{I})$ | $\boldsymbol{\varepsilon} \sim \mathcal{N}(\mathbf{0}, \sigma^2_{\varepsilon}\mathbf{I})$ | $\boldsymbol{\varepsilon} \sim \mathcal{N}(\mathbf{0}, \sigma^2_{\varepsilon}\mathbf{I})$ | $\boldsymbol{\varepsilon} \sim \mathcal{N}(\mathbf{0}, \sigma^2_{\varepsilon}\mathbf{I})$ | 다변량정규분포로 가정 |
| 랜덤효과벡터 분포 | — | — | $\mathbf{u} \sim \mathcal{N}(\mathbf{0}, \sigma^2_{u}\mathbf{I})$ | $\mathbf{a} \sim \mathcal{N}(\mathbf{0}, \sigma^2_{a}\mathbf{A})$ | $\quad \mathbf{g} \sim \mathcal{N}(\mathbf{0}, \sigma^2_{g}\mathbf{G})$ | $\mathbf{u} \sim \mathcal{N}(\mathbf{0}, \sigma^2_{u}\mathbf{H})$ | 다변량정규분포로 가정 |
| 랜덤효과 분포 | — | — | $\mathbf{Z}\mathbf{u} \sim \mathcal{N}(\mathbf{0}, \sigma_u^2\mathbf{ZZ}^\top )$ | $\mathbf{Z_a}\mathbf{a} \sim \mathcal{N}(\mathbf{0}, \sigma_a^2\mathbf{Z_aAZ_a}^\top)$ | $\mathbf{Z_g}\mathbf{g} \sim \mathcal{N}(\mathbf{0}, \sigma_g^2\mathbf{Z_gGZ_g}^\top)$ | $\mathbf{Z}\mathbf{u} \sim \mathcal{N}(\mathbf{0}, \sigma_u^2\mathbf{ZHZ}^\top)$ | 다변량정규분포로 변환 |
| 고정효과 | $\mathbf{X}\boldsymbol{\beta}$ | $\mathbf{X}\boldsymbol{\beta}$ | $\mu \mathbf{1}$ | $\mathbf{X}\boldsymbol{\beta}$ | $\mathbf{X}\boldsymbol{\beta}$ | $\mathbf{X}\boldsymbol{\beta}$ | 고정효과는 분포대신 제약조건 $\sum\limits_{i=1}^k\alpha_i=0$, $\mathbf{C}\boldsymbol{\beta} = \mathbf{0}, |
| $\mathbf{y}$의 분포 | $\mathbf{y} \sim \mathcal{N}(\mathbf{X}\boldsymbol{\beta}, \sigma_{\varepsilon}^2\mathbf{I})$ | $\mathbf{y} \sim \mathcal{N}(\mathbf{X}\boldsymbol{\beta}, \sigma_{\varepsilon}^2\mathbf{I})$ | $\mathbf{y} \sim \mathcal{N}( \mu \mathbf{1}, \sigma_u^2\mathbf{ZZ}^\top + \sigma_{\varepsilon}^2\mathbf{I})$ | $\mathbf{y} \sim \mathcal{N}(\mathbf{X}\boldsymbol{\beta}, \sigma_a^2\mathbf{Z_aAZ_a}^\top + \sigma_{\varepsilon}^2\mathbf{I})$ | $\mathbf{y} \sim \mathcal{N}(\mathbf{X}\boldsymbol{\beta}, \sigma_g^2\mathbf{Z_gGZ_g}^\top + \sigma_{\varepsilon}^2\mathbf{I})$ | $\mathbf{y} \sim \mathcal{N}(\mathbf{X}\boldsymbol{\beta}, \sigma_u^2\mathbf{ZHZ}^\top + \sigma_{\varepsilon}^2\mathbf{I})$ | 다변량정규분포로 변환 ANOVA $y_{ij} \sim \mathcal{N}(\mu + \alpha_i, \sigma_{\varepsilon}^2)$ |
| 고정효과벡터 | $\boldsymbol{\beta}$ | $\boldsymbol{\beta}$ | $\boldsymbol{\beta}$ | $\boldsymbol{\beta}$ | $\boldsymbol{\beta}$ | $\boldsymbol{\beta}$ | ANOVA는 평균차이 |
| 랜덤효과벡터 | — | — | $\mathbf{u}$ | $\mathbf{a}$ | $\mathbf{g}$ | $\mathbf{u}$ | $\mathbf{a}$, $\mathbf{a}$는 유전효과 |
| 관계행렬 | — | — | $\mathbf{I}$ | $\mathbf{A}$ | $\mathbf{G}$ | $\mathbf{H}$ | 개체 간에 서로 독립인 경우 $\mathbf{I}$ ssGBLUP는 $\mathbf{A}$가 $\mathbf{H}$를 반드시 포함 |
| 오차항의 분산성분 | $\sigma_{\varepsilon}^2$ | $\sigma_{\varepsilon}^2$ | $\sigma_{\varepsilon}^2$ | $\sigma_{\varepsilon}^2$ | $\sigma_{\varepsilon}^2$ | $\sigma_{\varepsilon}^2$ | |
| 랜덤효과벡터의 분산성분 | $\sigma_u^2$ | $\sigma_a^2$ | $\sigma_g^2$ | $\sigma_u^2$ | |||
| 오차항의 분산-공분산행렬 | $\operatorname{Var}(\boldsymbol{\varepsilon})=\sigma_{\varepsilon}^2\mathbf{I}$ | $\operatorname{Var}(\boldsymbol{\varepsilon})=\sigma_{\varepsilon}^2\mathbf{I}$ | $\operatorname{Var}(\boldsymbol{\varepsilon})=\sigma_{\varepsilon}^2\mathbf{I}$ | $\operatorname{Var}(\boldsymbol{\varepsilon})=\sigma_{\varepsilon}^2\mathbf{I}$ | $\operatorname{Var}(\boldsymbol{\varepsilon})=\sigma_{\varepsilon}^2\mathbf{I} $ | $\sigma_{\varepsilon}^2\mathbf{I}$ | 오차항은 모두 독립 등분산 가정 |
| 랜덤효과벡터의 분산-공분산행렬 | — | — | $\operatorname{Var}(\mathbf{u})=\sigma_u^2\mathbf{I} $ | $\operatorname{Var}(\mathbf{a})=\sigma_a^2\mathbf{A} $ | $\operatorname{Var}(\mathbf{g})=\sigma_g^2\mathbf{G} $ | $\operatorname{Var}(\mathbf{u})=\sigma_u^2\mathbf{H} $ | |
| 랜덤효과의 분산-공분산행렬 | — | — | $\operatorname{Var}(\mathbf{u})=\sigma_u^2\mathbf{ZZ}^\top $ | $\operatorname{Var}(\mathbf{a})=\sigma_a^2\mathbf{Z_aAZ_a}^\top $ | $\operatorname{Var}(\mathbf{g})=\sigma_g^2\mathbf{Z_gGZ_g}^\top $ | $\operatorname{Var}(\mathbf{u})=\sigma_u^2\mathbf{ZHZ}^\top $ | |
| 반응변수의 분산-공분산행렬 | $\operatorname{Var}(\mathbf{y})=\mathbf{V}=\sigma_{\varepsilon}^2\mathbf{I}$ | $\operatorname{Var}(\mathbf{y})=\mathbf{V}=\sigma_{\varepsilon}^2\mathbf{I}$ | $\operatorname{Var}(\mathbf{y})=\mathbf{V}=\sigma_u^2\mathbf{ZZ}^\top + \sigma_{\varepsilon}^2\mathbf{I}$ | $\operatorname{Var}(\mathbf{y})=\mathbf{V}=\sigma_a^2\mathbf{Z_aAZ_a}^\top + \sigma_{\varepsilon}^2\mathbf{I}$ | $\operatorname{Var}(\mathbf{y})=\mathbf{V}=\sigma_g^2\mathbf{Z_gGZ_g}^\top + \sigma_{\varepsilon}^2\mathbf{I}$ | $\operatorname{Var}(\mathbf{y})=\mathbf{V}=\sigma_u^2\mathbf{ZHZ}^\top \ + \sigma_{\varepsilon}^2\mathbf{I}$ |
Analysis of Variance
스칼라 모델식
$$\mathbf{y}_{ij} = \mu + \alpha_i + \varepsilon_{ij},\quad \varepsilon_{ij} \sim \mathcal{N}(0, \sigma^2)$$
여기서, $\mathbf{y}_{ij}$는 $i$번째 처리(또는 집단)에서 $j$번째 관측값 (총 $n$개)
$\mu$는 전체 평균 (모든 집단의 공통 평균)
$\alpha_i$는 $i$번째 집단(처리)의 효과 (처리 간 차이)
$\varepsilon_{ij}$는 오차항(정규분포, 평균 0, 분산 $\sigma^2$)
벡터 모델식 (1)
$$\mathbf{y} \sim \mathcal{N}(\mu \cdot \mathbf{1}_n + \boldsymbol{\alpha},\; \sigma^2 \mathbf{I})$$
여기서, $\mathbf{y}$는 관측값 전체 벡터 ($n\times1$)
$\mu \cdot \mathbf{1}_n$은 전체평균을 모든 관측값에 적용한 벡터
$\boldsymbol{\alpha}$는 처리 집단에 해당하는 효과 벡터 (예: 더미코딩 형태)
$\sigma^2 \mathbf{I}$는 등분산 정규오차 가정
벡터 모델식 (2)
$$\mathbf{y} = \mathbf{X}\boldsymbol{\beta} + \boldsymbol{\varepsilon} \quad , \quad \mathbf{y} \sim \mathcal{N}(\mathbf{X}\boldsymbol{\beta},\; \sigma^2 \mathbf{I})$$
여기서, $\mathbf{y} \in \mathbb{R}^{n \times 1}$는 관측값 벡터 (표현형 또는 종속변수)
$\mathbf{X} \in \mathbb{R}^{n \times p}$는 디자인 행렬 (독립변수, 설명변수)
$\boldsymbol{\beta} \in \mathbb{R}^{p \times 1}$는 고정효과 계수 벡터
$\sigma^2 \mathbf{I}$는 등분산 가정 하의 오차 공분산 행렬
$\mathcal{N}(\cdot,\cdot)$는 다변량 정규분포
추정하고자 하는 것은 모수
| 모수 기호 | 모수 명 |
|---|---|
| $\mu$ | 전체평균 |
| $\mu_i$ | $i$번째 집단평균 |
$\alpha_i$ $\alpha_i=\mu-\mu_i$ | 고정효과(fixed), 처리효과(treatment), 집단효과(group effect) 집단편차(group deviattion) |
| $\sigma^2$ | 오차의 분산 |
모수를 추정하는 추정량
| 추정량 기호 | 추정량 명 | 추정량 |
|---|---|---|
$\hat{\mu}$ ($\bar{y}_{\cdot\cdot}$) | 표본전체평균 | $$\hat{\mu} = \frac{1}{n} \sum_{i=1}^{k} \sum_{j=1}^{n_i} y_{ij}$$ 여기서, $n$은 표본크기 $k$는 표본내 집단수 |
$\hat{\mu}_i$ ($\bar{y}_{i\cdot}$) | 표본내 $i$번째 집단평균 | $$\hat{\mu} = \frac{1}{n} \sum_{i=1}^{k} \sum_{j=1}^{n_i} y_{ij}$$ 여기서, $n$은 표본크기 $k$는 표본내 집단수 |
$\hat{\mu}_i-\hat{\mu}$ ($\bar{y}_{i\cdot} – \bar{y}_{\cdot\cdot}$) | 표본내 $i$번째 집단평균 – 표본전체평균 | $$\hat{\mu}_i-\hat{\mu}=(\bar{y}_{i\cdot} – \bar{y}_{\cdot\cdot})=\alpha_i$$ |
| $\hat{\alpha}_i$ | 고정효과, 처리효과, 집단효과, 집단편차 | $$\hat{\alpha}_i =\frac{1}{n_i} \sum_{j=1}^{n_i} y_{ij} – \frac{1}{n} \sum_{i=1}^{k} \sum_{j=1}^{n_i} y_{ij}$$ |
| $\hat{\sigma}^2$ | 잔차의 분산 | $$\hat{\sigma}^2 = \frac{\sum\limits_{i=1}^{k} \sum\limits_{j=1}^{n_i} (y_{ij} – \hat{y}_{ij})^2}{n – k} = \frac{SS_E}{n – k}$$ $n_i$는 $i$번째 집단의 개체수 $n$은 전체 개체수 $k$는 집단수 |
최소제곱법(OLS, Ordinary Least Squares)으로 추정량 구하기
잔차제곱합($SS_E$, $SS_{Res}$)을 최소로 하는 $\hat{\mu}$, $\hat{\alpha_i}$ 찾기.
$$SS_E = \sum_{i=1}^{k} \sum_{j=1}^{n_i} \left( y_{ij} – \hat{\mu} – \hat{\alpha}_i \right)^2$$
단. 고유한 해를 갖기 위한 제약조건
$$\sum_{i=1}^{k} \hat{\alpha_i}= 0$$
여기서, $k$는 집단수
다음의 제약조건을 포함한 추정식인 라그랑주안 함수를 편미분하여 모평균 추정량 $\hat{\mu}$과 집단평균 추정량 $\hat{\alpha_i}$을 구함
$$\mathcal{L}(\mu, \alpha_1, \dots, \alpha_k, \lambda)= \sum_{i=1}^{k} \sum_{j=1}^{n_i} (y_{ij} – \mu – \alpha_i)^2+ \lambda \left( \sum_{i=1}^{a} \alpha_i \right)$$
추정값(최종해)를 계산하기 위한 전체평균 $\hat{\mu}$, 전체평균과 집단평균의 편차 $\hat{\alpha}_i$를 구한 결과
$$\hat{\mu} = \frac{1}{n} \sum_{i=1}^{k} \sum_{j=1}^{n_i} y_{ij}$$
$$\hat{\alpha}_i =\frac{1}{n_i} \sum_{j=1}^{n_i} y_{ij} – \frac{1}{N} \sum_{i=1}^{k} \sum_{j=1}^{n_i} y_{ij}$$
$\sigma^2$의 추정량 $\hat{\sigma}^2$을 구하면
$$\hat{\sigma}^2 = \frac{\sum\limits_{i=1}^{a} \sum\limits_{j=1}^{n_i} (y_{ij} – \hat{y}_{ij})^2}{n – k} = \frac{SS_E}{n – k}$$
가설의 유의성 검정
귀무가설
$$H_0: \alpha_1 = \alpha_2 = \cdots = \alpha_k = 0$$
대립가설
$$H_1: \text{적어도 하나의}\,\, \alpha_i \ne 0$$
검정통계량으로 사용하는 F통계량
$$F = \dfrac{MS_B}{MS_E} = \dfrac{\dfrac{SS_B}{k – 1}}{\dfrac{SS_E}{n – k}}$$
$\alpha_i$는 전체평균과 2021년, 2022년, 2023년 출생 한우의 근내지방도 평균의 차이
평균중심벡터 $\mathbf{\alpha}$
$$\boldsymbol{\alpha} = \mathbf{y} – \mu \cdot \mathbf{1}_n$$
여기서, $\mathbf{y}$는 $n\times 1$ 관측값벡터(근내지방도 %)
$\mu$는 전체평균(스칼라)
$\mathbf{1}_n$은 $n\times 1$인 원소가 모두 1인 벡터
$n$은 개체 수
$\mathbf{\alpha}$는 각 개체가 전체평균($\mu$)에서 얼마나 벗어났는지를 나타내는 벡터
평균중심벡터 $\mathbf{\alpha}$
$$\boldsymbol{\alpha} =
\begin{bmatrix}
\alpha_{2021} \\
\alpha_{2022} \\
\alpha_{2023} \\
\end{bmatrix}
$$
여기서, $\alpha_{2021}$는 2021년에 출생한 한우의 평균과 전체평균과의 차이
$\alpha_{2022}$는 2022년에 출생한 한우의 평균과 전체평균과의 차이
$\alpha_{2023}$는 2023년에 출생한 한우의 평균과 전체평균과의 차이
2021년을 기준범주(reference category)**로 설정하고 더미변수를 사용 $\mathbf{\alpha}$
$$\boldsymbol{\alpha} =
\begin{bmatrix}
0 \\
\alpha_{2022} \\
\alpha_{2023} \\
\end{bmatrix}
\quad \text{(with 2021 as reference)}
$$
여기서, $\alpha_{2022}$는 2021년에 출생한 한우의 평균과 2021년에 출생한 한우의 평균의 차이
$\alpha_{2023}$는 2021년에 출생한 한우의 평균과 2023년에 출생한 한우의 평균의 차이
Fisher (1925), 유전학자, 통계학의 시조
처리 효과와 오차 분해. 선형모형의 출발점
시험육종, 품종비교실험
실험설계, 생물학, 약리학
Best Linear Unbiased Estimator
벡터 모델식
$$\mathbf{y} = \mathbf{X}\boldsymbol{\beta} + \boldsymbol{\varepsilon} \quad , \quad \mathbf{y} \sim \mathcal{N}(\mathbf{X}\boldsymbol{\beta},\; \sigma^2 \mathbf{I})$$
여기서, $\mathbf{y} \in \mathbb{R}^{n \times 1}$는 관측값 벡터 (표현형 또는 종속변수)
$\mathbf{X} \in \mathbb{R}^{n \times p}$는 디자인 행렬 (독립변수, 설명변수)
$\boldsymbol{\beta} \in \mathbb{R}^{p \times 1}$는 고정효과 계수 벡터
$\sigma^2 \mathbf{I}$는 등분산 가정 하의 오차 공분산 행렬
$\mathcal{N}(\cdot,\cdot)$는 다변량 정규분포
다변량 정규분포를 나타내는 벡터와 그 벡터의 선형변환
오차항: $\boldsymbol{\varepsilon}$
$$\boldsymbol{\varepsilon}$$
$$\boldsymbol{\varepsilon} \sim \mathcal{N}(0, \sigma_{\varepsilon}^2\mathbf{I})$$
반응변량: $\mathbf{y}$
$$\mathbf{y}=\mathbf{X\beta}+\boldsymbol{\varepsilon}$$
$$\mathbf{y} \sim \mathcal{N}(\mathbf{X}\boldsymbol{\beta}, \sigma^2 \mathbf{I})$$
BLUE: $\hat{\mathbf{\beta}}$
$$\hat{\boldsymbol{\beta}} = (\mathbf{X}^\top \mathbf{X})^{-1} \mathbf{X}^\top \mathbf{y}$$
$$\hat{\boldsymbol{\beta}} \sim \mathcal{N} \left( \boldsymbol{\beta},\ \sigma^2 (\mathbf{X}^\top \mathbf{X})^{-1} \right)$$
| 용어 | 설명 | 의미 |
|---|---|---|
| Best | 최선의 (최소 분산) | 동일한 조건의 다른 추정량보다 분산이 가장 작다 |
| Linear | 선형 | 추정량 $\hat{\boldsymbol{\beta}}$은 관측값 $\mathbf{y}$의 선형결합으로 표현된다. $\hat{\boldsymbol{\beta}} = (\mathbf{X}^\top \mathbf{X})^{-1} \mathbf{X}^\top \mathbf{y}$ 여기서, $\mathbf{X}$은 입력데이터로 상수이므로 $(\mathbf{X}^\top \mathbf{X})^{-1} \mathbf{X}^\top$도 고정행렬(비확률적 상수) |
| Unbiased | 불편 | 기대값이 진짜 모수와 같다: $\mathbb{E}[\hat{\boldsymbol{\beta}}] = \boldsymbol{\beta}$ |
| Estimator | 추정량 | 모수를 추정하는 식, 보통 표본에서 계산됨 |
모수와 모수를 추정하는 방법과 추정량
| 모수 | 추정 방 | 추정량 |
|---|---|---|
회귀계수벡터(고정효과벡터) $\boldsymbol{\beta}$ | 최소제곱법 또는 최대우도법 | $\hat{\boldsymbol{\beta}} = (\mathbf{X}^\top \mathbf{X})^{-1} \mathbf{X}^\top \mathbf{y}$ |
오차 분산 $\sigma^2$ | 잔차 제곱합을 자유도로 나눔 | $\hat{\sigma}^2 = \dfrac{\mathbf{e}^\top \mathbf{e}}{n – p}$ 여기서, $n$은 표본크기 $p$는 표본내집단수 |
최소제곱법(OLS, Ordinary Least Square)으로 $\boldsymbol{\beta}$를 추정
최소제곱법으로 구하는 추정량은 잔차제곱합을 최소화하는 목적함수로부터 유도되며, 그 해는 선형회귀계수의 추정량이며 Gauss-Markov 가정 하에서 모든 선형불편추정량 중 평균제곱오차(MSE)가 최소가 되는 최선선형불편추정량(BLUE)임. 오차항이 정규분포가 아니더라도 모델이 선형성 ($\boldsymbol{X}\beta$가 상수)이 있고 오차의 기대값이 0이고 분산이 등분산이고 독립성이 있으면 최소제곱추정량은 모든 선형불편추정량 중에서 분산이 가장 작은 최적추정량 BLUE(Best Linear Unbiased Estimator)임. 이를 Gauss-Markov 정리라고 함.
잔차제곱합($SS_{Res}$, $SS_E$)을 최소화하는 $\boldsymbol{\beta}$를 구하고 이를 $\hat{\boldsymbol{\beta}}$이라 함.
$$SS_{Res}=\sum_{i=1}^{n} (y_i – \hat{y}_i)^2 = (\mathbf{y} – \mathbf{X}\boldsymbol{\beta})^\top (\mathbf{y} – \mathbf{X}\boldsymbol{\beta})=\mathbf{e}^\top \mathbf{e}$$
위의 추정식의 미분값이 0이 되는 조건에서의 최소제곱 추정량 $\hat{\boldsymbol{\beta}}_{\text{OLS}}$을 구함. 단 $\mathbf{X}^\top \mathbf{X}$가 가역(invertible)적이어야 고유해를 구할 수 있음.
$$\hat{\boldsymbol{\beta}}_{\text{OLS}} = (\mathbf{X}^\top \mathbf{X})^{-1} \mathbf{X}^\top \mathbf{y}$$
증명
고정효과 선형모델 BLUE의 데이터를 설명하는 회귀모델은 다음과 같은 수식으로 표현.
$$\mathbf{y} = \mathbf{X} \boldsymbol{\beta} + \mathbf{e}$$
여기서 $\boldsymbol{\beta}$는 회귀계수 벡터이고, $\mathbf{e}$는 오차벡터.
오차의 제곱합을 최소화하는 $\boldsymbol{\beta}$를 찾아야 함.
$$SS_{Res}=SS_E=S(\boldsymbol{\beta}) = \| \mathbf{y} – \mathbf{X} \boldsymbol{\beta} \|^2 = (\mathbf{y} – \mathbf{X} \boldsymbol{\beta})^T (\mathbf{y} – \mathbf{X} \boldsymbol{\beta})$$
위 함수를 전개하면,
$$S(\boldsymbol{\beta}) = \mathbf{y}^T \mathbf{y} – 2 \boldsymbol{\beta}^T \mathbf{X}^T \mathbf{y} + \boldsymbol{\beta}^T \mathbf{X}^T \mathbf{X} \boldsymbol{\beta}$$
위 함수의 최소값을 찾기 위해 $\boldsymbol{\beta}$에 대해 미분.
$$\frac{\partial S(\boldsymbol{\beta})}{\partial \boldsymbol{\beta}} = -2 \mathbf{X}^T \mathbf{y} + 2 \mathbf{X}^T \mathbf{X} \boldsymbol{\beta}$$
최소값에서 기울기는 0이므로, 위 식을 0으로 놓음.
$$-2 \mathbf{X}^T \mathbf{y} + 2 \mathbf{X}^T \mathbf{X} \boldsymbol{\beta} = 0$$
양변을 2로 나누고 정리하면:
$$\mathbf{X}^T \mathbf{X} \boldsymbol{\beta} = \mathbf{X}^T \mathbf{y}$$
만약, $\mathbf{X}^T \mathbf{X}$가 역행렬을 가질 수 있다면 (정칙행렬이라면),
$$\boldsymbol{\beta} = (\mathbf{X}^T \mathbf{X})^{-1} \mathbf{X}^T \mathbf{y}$$
최대우도법(ML, Maximum Likelihood)으로 $\boldsymbol{\beta}$ 추정
다변량 정규분포를 가지는 확률변수벡터 $\mathbf{y}$의 결합확률밀도함수는
$$f(\mathbf{y} \mid \boldsymbol{\beta}, \sigma^2)
= \frac{1}{(2\pi\sigma^2)^{n/2}} \exp\left( -\frac{1}{2\sigma^2} (\mathbf{y} – \mathbf{X}\boldsymbol{\beta})^\top (\mathbf{y} – \mathbf{X}\boldsymbol{\beta}) \right)$$
위의 결합밀도함수의 우도함수에 로그를 취한 로그우도함수는
$$\log L(\boldsymbol{\beta}, \sigma^2 \mid \mathbf{y})
= -\frac{n}{2} \log(2\pi\sigma^2)
– \frac{1}{2\sigma^2} (\mathbf{y} – \mathbf{X}\boldsymbol{\beta})^\top (\mathbf{y} – \mathbf{X}\boldsymbol{\beta})$$
로그우도함수로부터 최대우도추정량(MLE, Maximum Likelihood Estimator) $\hat{\boldsymbol{\beta}}_{\text{MLE}}$을 구하면
$$\hat{\boldsymbol{\beta}}_{\text{MLE}} = (\mathbf{X}^\top \mathbf{X})^{-1} \mathbf{X}^\top \mathbf{y}$$
| 오차항 조건 | 회귀계수벡터 $\boldsymbol{\beta}$의 추정량 기호와 추정량 | 오차항 조건에 따른 추정량 설명 |
|---|---|---|
| 정규성 없음 | $\hat{\boldsymbol{\beta}}_{\text{OLS}} = (\mathbf{X}^\top \mathbf{X})^{-1} \mathbf{X}^\top \mathbf{y}$ | 오차항이 정규분포를 따르지 않아도, Gauss–Markov 조건만 만족하면 OLS로 추정한 $\hat{\boldsymbol{\beta}}_{\text{OLS}}$는 최선선형불편추정량 (BLUE, Best Linear Unbiased Estimaor)임 |
| 정규성 있음 | $\hat{\boldsymbol{\beta}}_{\text{MLE}}=(\mathbf{X}^\top \mathbf{X})^{-1} \mathbf{X}^\top \mathbf{y}$ 여기서, $\hat{\boldsymbol{\beta}}_{\text{MLE}}=\hat{\boldsymbol{\beta}}_{\text{OLS}}$ | 오차항을 등분산 다변량 정규분포로 가정하면 $$\boldsymbol{\varepsilon} \sim \mathcal{N}(0, \sigma^2 \mathbf{I})$$ 선형변환에 따라 선형모델도 다변량 정규분포 $$\mathbf{y} = \mathbf{X} \boldsymbol{\beta} + \boldsymbol{\varepsilon}$$ 여기서, $\mathbf{X} \boldsymbol{\beta}$는 상수항 $\mathbf{y} \sim \mathcal{N}(\mathbf{X} \boldsymbol{\beta}, \sigma^2 \mathbf{I})$ $\mathbf{y}$의 확률밀도함수 $f(\boldsymbol{y}|\boldsymbol{\beta},\sigma^2)$가 존재하고 로그우드함수 $\log L(\boldsymbol{\beta}, \sigma^2 \mid \mathbf{y})$를 정의할 수 있음. 따라서 최대우도추정량(MLE)를 구할 수 있고 OLE로 구한 추정량과 같음, 그리고 두 추정량은 모두 BLUE임 |
오차분산 $\sigma^2$의 추정량: 자유도를 적용한 불편추정량(Unbiased Estimator)
오차분산의 추정량
$$\hat{\sigma}^2 = \frac{\mathbf{e}^\top \mathbf{e}}{n – p}$$
여기서, $y_i$는 관측값
$\hat{y}_i$는 예측값
$\mathbf{e} = \mathbf{y} – \mathbf{X} \hat{\boldsymbol{\beta}}$는 잔차벡터
$\mathbf{e}^\top \mathbf{e}$는 잔차제곱합의 벡터 표현
$n$은 관측값 개수
$p$는 추정된 계수의 수 (설계행렬 $\boldsymbol{X}$의 열 수)
$\mathbf{y}$: 반응변량의 관측값 벡터 (근내지방도)
절편을 가지는 설계행렬 $\mathbf{X}$ 구성
$$\mathbf{X} =
\begin{bmatrix}
1 & 0 & 0 & 435.2 \\
1 & 1 & 0 & 420.7 \\
1 & 0 & 1 & 462.5 \\
1 & 0 & 0 & 448.0 \\
1 & 1 & 0 & 410.3 \\
1 & 0 & 1 & 472.6 \\
1 & 0 & 0 & 437.8 \\
1 & 1 & 0 & 455.4 \\
\end{bmatrix}$$
계수벡터 $\boldsymbol{\beta}$
$$\boldsymbol{\beta} =
\begin{bmatrix}
\beta_0 \\
\beta_1 \\
\beta_2 \\
\beta_3 \\
\end{bmatrix}
$$
여기서, $\beta_0$는 절편(intercept)
$\beta_1$는 BirthYear_2022 (2022 = 1, else 0)
$\beta_2$는 BirthYear_2023 (2023 = 1, else 0)
$\beta_3$는 CarcassWeight (도체중, kg)
Aitken (1935), 수리통계학자
고정효과의 최적 선형 추정량
형질간 회귀 분석
경제학, 회귀분석, 기업 성과 예측
Best Linear Unbiased Prediction
벡터 모덿식
$$$\mathbf{y} = \mu \mathbf{1} + \mathbf{Z}\mathbf{u} + \boldsymbol{\varepsilon} \quad , \quad \mathbf{y} \sim \mathcal{N}(\mu \mathbf{1},\; \sigma_u^2\mathbf{ZZ}^\top + \sigma_{\varepsilon}^2\mathbf{I})$$
여기서, $\mathbf{y} \in \mathbb{R}^{n \times 1}$는 표현형(관측값) 벡터
$ \mu \mathbf{1}$는 고정효과
$\mathbf{Z} \in \mathbb{R}^{n \times n}$는 랜덤효과 설계행렬
$\sigma^2_u$는 랜덤효과벡터의 분산성분
$\sigma^2_{\epsilon}$은 오차항 분산성분
$\mathbf{I}$는 항등행렬
다변량 정규분포를 나타내는 벡터와 그 벡터의 선형변환
오차항: $\boldsymbol{\varepsilon}$
$$\boldsymbol{\varepsilon}$$
$$\boldsymbol{\varepsilon} \sim \mathcal{N}(0, \sigma_{\varepsilon}^2\mathbf{I})$$
랜덤효과 벡터: $\mathbf{u}$
$$\mathbf{u}$$
$$\mathbf{u} \sim \mathcal{N}(0, \sigma_u^2\mathbf{G})$$
반응변량: $\mathbf{y}$
$$\mathbf{y} = \mu\boldsymbol{1} + \mathbf{Z}\mathbf{u} + \boldsymbol{\varepsilon}$$
$$\mathbf{y} \sim \mathcal{N}(\mathbf{X}\boldsymbol{\beta},\ \sigma^2_u \mathbf{ZGZ}^\top + \sigma^2_\varepsilon \mathbf{I})$$
BLUE: $\hat{\boldsymbol{\beta}}$
$$\hat{\boldsymbol{\beta}} = (\mathbf{X}^\top \mathbf{X})^{-1} \mathbf{X}^\top \mathbf{y}$$
$$\hat{\boldsymbol{\beta}} \sim \mathcal{N} \left( \boldsymbol{\beta},\ \sigma^2 (\mathbf{X}^\top \mathbf{X})^{-1} \right)$$
BLUP: $\hat{\mathbf{u}}$
$$\hat{\mathbf{u}} = \sigma^2_u \mathbf{G} \mathbf{Z}^\top \mathbf{V}^{-1} (\mathbf{y} – \mathbf{X} \hat{\boldsymbol{\beta}})$$
$$\hat{\mathbf{u}} \sim \mathcal{N} \left( \mathbf{a},\ \mathbf{CVC}^\top \right)$$
여기서, $\mathbf{C} = \sigma_u^2 \mathbf{G} \mathbf{Z}^\top \mathbf{V}^{-1} \left( \mathbf{I} – \mathbf{X}(\mathbf{X}^\top \mathbf{V}^{-1} \mathbf{X})^{-1} \mathbf{X}^\top \mathbf{V}^{-1} \right)$
| 용어 | 설명 | 의미 |
|---|---|---|
| Pedigree-based | 혈통 기반 | 예측 시 개체 간 유전적 유사도는 혈 정보를 기반으로 한 관계행렬 A를 사용함 |
| Best | 최선의 (최소 분산) | 동일한 조건의 다른 예측량보다 분산이 가장 작음. |
| Linear | 선형 |
예측량 $\hat{u}$는 관측값 $\mathbf{y}$의 선형결합으로 표현됨 $$ \begin{bmatrix} \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{X} & \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{Z} \\ \mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{X} & \mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{Z} + \mathbf{G}^{-1} \end{bmatrix} \begin{bmatrix} \hat{\boldsymbol{\beta}} \\ \hat{\mathbf{u}} \end{bmatrix} = \begin{bmatrix} \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{y} \\ \mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{y} \end{bmatrix} $$ |
| Unbiased | 불편 | 기댓값이 진짜 육종가와 같음. $\mathbb{E}[\hat{u}] = u$ |
| Prediction | 예측량 | 혈도 정보를 바탕으로 개체의 유전적 가치를 예측한 값 |
다변량 정규분포를 나타내는 벡터와 그 벡터의 선형변환
오차항: $\boldsymbol{\varepsilon}$
$$\boldsymbol{\varepsilon}$$
$$\boldsymbol{\varepsilon} \sim \mathcal{N}(0, \sigma_{\varepsilon}^2\mathbf{I})$$
확률효과 벡터: $\mathbf{g}$
$$\mathbf{g}$$
$$\mathbf{g} \sim \mathcal{N}(0, \mathbf{G}\sigma_g^2)$$
반응변량: $\mathbf{y}$
$$\mathbf{y} = \mathbf{X}\boldsymbol{\beta} + \mathbf{Z_g}\mathbf{g} + \boldsymbol{\varepsilon}$$
$$\mathbf{y} \sim \mathcal{N}(\mathbf{X}\boldsymbol{\beta},\ \sigma^2_g \mathbf{Z_gGZ_g}^\top + \sigma^2_\varepsilon \mathbf{I})$$
BLUE: $\hat{\boldsymbol{\beta}}$
$$\hat{\boldsymbol{\beta}} = (\mathbf{X}^\top \mathbf{X})^{-1} \mathbf{X}^\top \mathbf{y}$$
$$\hat{\boldsymbol{\beta}} \sim \mathcal{N} \left( \boldsymbol{\beta},\ \sigma^2 (\mathbf{X}^\top \mathbf{X})^{-1} \right)$$
BLUP: $\hat{\mathbf{g}}$
$$\hat{\mathbf{g}} = \sigma^2_g \mathbf{G} \mathbf{Z_g}^\top \mathbf{V_g}^{-1} (\mathbf{y} – \mathbf{X} \hat{\boldsymbol{\beta}})$$
$$\hat{\mathbf{g}} \sim \mathcal{N} \left( \mathbf{g},\ \mathbf{C_gV_gC_g}^\top \right)$$
여기서, $\mathbf{C_g} = \sigma_g^2 \mathbf{A} \mathbf{Z_g}^\top \mathbf{V_g}^{-1} \left( \mathbf{I} – \mathbf{X}(\mathbf{X}^\top \mathbf{V_g}^{-1} \mathbf{X})^{-1} \mathbf{X}^\top \mathbf{V_g}^{-1} \right)$
모수와 모수를 추정하는 방법과 추정량
BLUP는 MME를 통해 고정효과와 확률효과를 동시에 추정
분산성분은 REML 등으로 추정
| 모수 | 추정방법 | 추정량 |
|---|---|---|
| 회귀계수벡터 (고정효과벡터) $\boldsymbol{\beta}$ | MME 이용한 고정효과 추정 | $\hat{\boldsymbol{\beta}} = (\mathbf{X}^\top \mathbf{X})^{-1}\mathbf{X}^\top(\mathbf{y}-\mathbf{Z}\hat{\mathbf{u}})$ |
| 확률효과벡터 (개체효과) $\mathbf{u}$ | MME 이용한 확률효과 추정 | $\hat{\mathbf{u}} = (\mathbf{Z}^\top \mathbf{Z}+\lambda \mathbf{I})^{-1}\mathbf{Z}^\top(\mathbf{y}-\mathbf{X}\hat{\boldsymbol{\beta}})$ |
| 오차항 분산성분 $\sigma_\varepsilon^2$ | REML 또는 잔차제곱합 기반 추정 | $\hat{\sigma}_\varepsilon^2 = \dfrac{\hat{\mathbf{e}}^\top\hat{\mathbf{e}}}{\,n-p\,}$ |
| 확률효과 분산성분 $\sigma_u^2$ | REML 반복 알고리즘 (예: EM, AI) |
모델
$$\mathbf{y} = \mathbf{X}\boldsymbol{\beta} + \mathbf{Z}\mathbf{u} + \boldsymbol{\varepsilon}$$
여기서, $\mathbf{y} \sim \mathcal{N}(\mathbf{X}\boldsymbol{\beta},\, \mathbf{V})$
$\mathbf{V}$는 $\mathbf{y}$의 공분산행렬: $\mathbf{V} = \mathbf{Z}\mathbf{Z}^\top + \mathbf{R}$
$\mathbf{u}$는 랜덤효과의 벡터: $\mathbf{u} \sim \mathcal{N}(\boldsymbol{0}, \sigma_u^2\mathbf{I})$
$\mathbf{R}$은 오차항(잔차)의 공분산행렬: $\mathbf{R} = \sigma_\varepsilon^2 \mathbf{I}$
랜덤효과 선형모델은 최소제곱법만으로는 모수의 추정과 확률변수값의 예측을 적절히 수행할 수 없으며, 혼합모델 해법(BLUP)과 분산 추정을 위한 MLE/REML이 모두 필요. 특히 REML + BLUP 조합이 가장 흔하게 사용됨.
목적함수를 유도하고 이를 미분하여 함수값의 최소조건으로 혼합모델방정식(MME, Mixed Model Equation)을 도출
목적함수 $Q(\boldsymbol{\beta}, \,\mathbf{u})$의 유도
1. 선형혼합모델
$$\mathbf{y} = \mu \boldsymbol{1} + \mathbf{Z} \mathbf{u} + \boldsymbol{\varepsilon}$$
여기서, $\mathbf{u} \sim \mathcal{N}(0, \sigma_u^2\mathbf{I})$
$\boldsymbol{\varepsilon} \sim \mathcal{N}(0, \sigma_{\varepsilon}^2\mathbf{I})$
2. $\mathbf{y}$의 조건부분포
$$\mathbf{y} \mid \mathbf{u} \sim \mathcal{N}(\mu \boldsymbol{1} + \mathbf{Z} \mathbf{u}, \sigma_{\varepsilon}^2\mathbf{I})$$
3. $\mathbf{u}$의 사전분포
$$ \mathbf{a} \sim \mathcal{N}(0, \sigma_u^2\mathbf{I})$$
4. 결합우도(joint likelihood)
$$f(\mathbf{y}, \mathbf{u} \mid \mu\boldsymbol{1}) = f(\mathbf{y} \mid \mathbf{u}, \mu\boldsymbol{1}) \cdot f(\mathbf{u})$$
5. 결합우도에 로그를 취하면
$$\log L
= -\tfrac{1}{2} (\mathbf{y} – \mu \boldsymbol{1} – \mathbf{Z}\mathbf{u})^\top
(\sigma_\varepsilon^2 \mathbf{I})^{-1}
(\mathbf{y} – \mu \boldsymbol{1} – \mathbf{Z}\mathbf{u})
– \tfrac{1}{2} \mathbf{u}^\top (\sigma_u^2 \mathbf{I})^{-1} \mathbf{u}
+ \text{const}$$
6. 목적함수 $Q(\boldsymbol{\mu}, \,\mathbf{u})$로 정리
$$Q(\mu, \mathbf{u}) =
(\mathbf{y} – \mu \boldsymbol{1} – \mathbf{Z}\mathbf{u})^\top
(\sigma_\varepsilon^2 \mathbf{I})^{-1}
(\mathbf{y} – \mu \boldsymbol{1} – \mathbf{Z}\mathbf{u})
+ \mathbf{u}^\top (\sigma_u^2 \mathbf{I})^{-1} \mathbf{u}$$
목적함수 $Q(\boldsymbol{\mu}, \,\mathbf{u})$의 앞쪽 항은 오차항의 가중제곱합, 뒤쪽 항은 랜덤효과의 정규분포 페널티. 이 목적함수를 미분하여 MME를 유도. MME(혼합모델방정식, Mixed Model Equation)는 고정효과와 랜덤효과를 동시에 추정하기 위한 시스템. 선형 모델과 정규분포 가정, 선형최소제곱 또는 우도최적화로부터 유도됨
목적함수(Q)를 편미분하여 0으로 설정
$\mu$에 대해 편미분
$$\boldsymbol{1}^\top \mathbf{R}^{-1} \boldsymbol{1} \,\mu
+ \boldsymbol{1}^\top \mathbf{R}^{-1} \mathbf{Z}\mathbf{u}
= \boldsymbol{1}^\top \mathbf{R}^{-1} \mathbf{y}$$
$\mathbf{u}$로 편미분
$$\mathbf{Z}^\top \mathbf{R}^{-1} \boldsymbol{1}\,\mu
+ (\mathbf{Z}^\top \mathbf{R}^{-1}\mathbf{Z} + \mathbf{G}^{-1})\mathbf{u}
= \mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{y}$$
편미분한 두 식을 행렬로 정리 -> MME 도출
MME(혼합모델방정식, Mixed Model Equation)는 다음식으로 표현됨
$$
\begin{bmatrix}
\boldsymbol{1}^\top \mathbf{R}^{-1} \boldsymbol{1} & \boldsymbol{1}^\top \mathbf{R}^{-1} \mathbf{Z} \\
\mathbf{Z}^\top \mathbf{R}^{-1} \boldsymbol{1} & \mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{Z} + \mathbf{G}^{-1}
\end{bmatrix}
\begin{bmatrix}
\hat{\mu} \\
\hat{\mathbf{u}}
\end{bmatrix}
=
\begin{bmatrix}
\boldsymbol{1}^\top \mathbf{R}^{-1} \mathbf{y} \\
\mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{y}
\end{bmatrix}
$$
여기서, $\mathbf{u} \sim \mathcal{N}(\sigma_u^2 \mathbf{I})$
REML(제한최대우도, Restricted Maximum Likelihood) 추정 과정
BLUP에서의 분산행렬($\mathbf{G}$, $\mathbf{R}$)과 분산성분($\sigma_u^2$, $\sigma_{\varepsilon}^2$)
$$\mathbf{G} = \sigma_u^2 \mathbf{I} \quad , \quad \mathbf{R} = \sigma_{varepsilon}^2 \mathbf{I}$$
분산성분, 회귀계수벡터, 육종가벡터를 REML으로 추정하는 과정
분산성분 수렴기준(convergence criterion)
| 알고리즘 | 수렴 기준 | 일반 값 |
|---|---|---|
| EM-REML | 절대 변화량 or 상대 변화량 | $10^{-6}$ |
| AI-REML | 로그우도 변화 or 분산성분 상대오차 | $10^{-8}$ |
| 소프트웨어 | asreml, sommer, lme4, blupf90 등 | 기본값 또는 설정 가능 |
모델 평가
| 평가 방법 | 내용 |
|---|---|
| 상관계수 | $\mathrm{cor}(\mathbf{\hat{u}, y}) / \sqrt{h^2}$ |
| MSE | $\frac{1}{n} \sum (y_i – \hat{y}_i)^2$ |
| LRT | $\sigma_u^2$ 유의성 검정 |
| AIC/BIC | 모형 비교용 |
| Cross-validation | 외부 데이터로 검증 정확도 평가 |
y : 반응변수id : 랜덤효과 레벨 (예: 개체ID)
Henderson (1975), 유전육종학자
단위행렬 기반 유전효과 예측
개체 간 독립성을 가정하고, 모든 랜덤효과가 동일한 분산을 가진다고 설정
잔차와 개체효과의 분산을 분리하여 추정
– 가축·작물 육종, 개량
– 계층형 조직 분석, 인사 평가
– 기업가치 요인 추정, 사회·생물학적 자료 분석
Pedigree-Based Best Linear Unbiased Prediction
벡터 모덿식
$$\mathbf{y} = \mathbf{X}\boldsymbol{\beta} + \mathbf{Z_a}\mathbf{a} + \boldsymbol{\varepsilon} \quad , \quad \mathbf{y} \sim \mathcal{N}(\mathbf{X}\boldsymbol{\beta},\; \mathbf{Z_aAZ_a}^\top \sigma_a^2 + \mathbf{I}\sigma_e^2)$$
여기서, $\mathbf{y} \in \mathbb{R}^{n \times 1}$는 표현형(관측값) 벡터
$\mathbf{X} \in \mathbb{R}^{n \times p}$는 고정효과 디자인 행렬
$\boldsymbol{\beta} \in \mathbb{R}^{p \times 1}$는 고정효과 계수
$\mathbf{Z_a} \in \mathbb{R}^{n \times q}$는 개체의 유전효과 할당 행렬 (통상 단위행렬 비슷한 형태)
$\mathbf{A} \in \mathbb{R}^{q \times q}$는 혈연관계행렬 (pedigree relationship matrix)
$\sigma^2_a$는 유전(우성) 효과의 분산
$\sigma^2_e$는 환경 오차의 분산
$\mathbf{I}$는 항등행렬 (단순한 오차 구조)
다변량 정규분포를 나타내는 벡터와 그 벡터의 선형변환
오차항: $\boldsymbol{\varepsilon}$
$$\boldsymbol{\varepsilon}$$
$$\boldsymbol{\varepsilon} \sim \mathcal{N}(0, \sigma_{\varepsilon}^2\mathbf{I})$$
랜덤효과 벡터: $\mathbf{a}$
$$\mathbf{a}$$
$$\mathbf{a} \sim \mathcal{N}(0, \sigma_a^2\mathbf{A})$$
반응변량: $\mathbf{y}$
$$\mathbf{y} = \mathbf{X}\boldsymbol{\beta} + \mathbf{Z_a}\mathbf{a} + \boldsymbol{\varepsilon}$$
$$\mathbf{y} \sim \mathcal{N}(\mathbf{X}\boldsymbol{\beta},\ \sigma^2_a \mathbf{Z_aAZ_a}^\top + \sigma^2_\varepsilon \mathbf{I})$$
BLUE: $\hat{\boldsymbol{\beta}}$
$$\hat{\boldsymbol{\beta}} = (\mathbf{X}^\top \mathbf{X})^{-1} \mathbf{X}^\top \mathbf{y}$$
$$\hat{\boldsymbol{\beta}} \sim \mathcal{N} \left( \boldsymbol{\beta},\ \sigma^2 (\mathbf{X}^\top \mathbf{X})^{-1} \right)$$
BLUP: $\hat{\mathbf{a}}$
$$\hat{\mathbf{a}} = \sigma^2_a \mathbf{A} \mathbf{Z_a}^\top \mathbf{V_a}^{-1} (\mathbf{y} – \mathbf{X} \hat{\boldsymbol{\beta}})$$
$$\hat{\mathbf{a}} \sim \mathcal{N} \left( \mathbf{a},\ \mathbf{C_aV_aC_a}^\top \right)$$
여기서, $\mathbf{C_a} = \sigma_a^2 \mathbf{A} \mathbf{Z_a}^\top \mathbf{V}^{-1} \left( \mathbf{I} – \mathbf{X}(\mathbf{X}^\top \mathbf{V}^{-1} \mathbf{X})^{-1} \mathbf{X}^\top \mathbf{V}^{-1} \right)$
| 용어 | 설명 | 의미 |
|---|---|---|
| Pedigree-based | 혈통 기반 | 예측 시 개체 간 유전적 유사도는 혈 정보를 기반으로 한 관계행렬 A를 사용함 |
| Best | 최선의 (최소 분산) | 동일한 조건의 다른 예측량보다 분산이 가장 작음. |
| Linear | 선형 |
예측량 $\hat{u}$는 관측값 $\mathbf{y}$의 선형결합으로 표현됨 $$ \begin{bmatrix} \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{X} & \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{Z} \\ \mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{X} & \mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{Z} + \mathbf{G}^{-1} \end{bmatrix} \begin{bmatrix} \hat{\boldsymbol{\beta}} \\ \hat{\mathbf{u}} \end{bmatrix} = \begin{bmatrix} \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{y} \\ \mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{y} \end{bmatrix} $$ |
| Unbiased | 불편 | 기댓값이 진짜 육종가와 같음. $\mathbb{E}[\hat{u}] = u$ |
| Prediction | 예측량 | 혈도 정보를 바탕으로 개체의 유전적 가치를 예측한 값 |
모수와 모수를 추정하는 방법과 추정량
PBLUP은 MME를 통해 고정효과와 확률효과를 동시에 추정
분산성분은 REML 등으로 추정
| 모수 | 추정방법 | 추정량 |
|---|---|---|
회귀계수벡터 (고정효과벡터) $\boldsymbol{\beta}$ | 혼합모델방정식(MME) 이용한 고정효과 추정 | $\hat{\boldsymbol{\beta}} = \left( \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{X} \right)^{-1} \mathbf{X}^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{Z_a} \hat{\mathbf{a}})$ |
혈통 육종가벡터 (확률효과벡터) $\mathbf{a}$ | 혼합모델방정식(MME) 이용한확률효과 추정 | $\hat{\mathbf{a}} = \left( \mathbf{Z_a}^\top \mathbf{R}^{-1} \mathbf{Z_a} + \mathbf{G_a}^{-1} \right)^{-1} \mathbf{Z_a}^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{X} \hat{\boldsymbol{\beta}})$ 여기서, $\mathbf{G_a} = \sigma_a^2 \mathbf{A}$ |
오차항 분산성분 $\sigma_{\varepsilon}^2$ | REML 또는 잔차제곱합 기반 추정 | $\hat{\sigma}_e^2 = \dfrac{\mathbf{e}^\top \mathbf{e}}{n – p}$ 여기서, $n$은 관측값 개수 $p$는 고정효과의 수 |
육종가벡터 분산성분 $\sigma_a^2$ | REML로 분산성분 추정 | 추정량은 없고 최대우도 기반 반복 알고리즘 사용 (예: EM-REML) |
모델
$$\mathbf{y} = \mathbf{X} \boldsymbol{\beta} + \mathbf{Z_a} \mathbf{a} + \boldsymbol{\varepsilon}$$
여기서, $\mathbf{y} \sim N(\mathbf{X} \boldsymbol{\beta}, \mathbf{V_a})$
$\mathbf{V_a}$는 $\mathbf{y}$의 공분산행렬: $\mathbf{V_a}=\mathbf{Z_a} \mathbf{G_a} \mathbf{Z_a}^\top + \mathbf{R}$
$\mathbf{G_a}$는 혈통 육종가의 공분산행렬: $\mathbf{G_a}=\sigma_a^2\mathbf{A}$
$\mathbf{R}$은 오차항(잔차)의 공분산행렬: $\mathbf{R} = \sigma_\varepsilon^2 \mathbf{I}$
PBLUP는 최소제곱법 만으로는 적절히 수행할 수 없으며, 혼합모형 해법(BLUP)과 분산 추정을 위한 MLE/REML이 모두 필요. 특히 REML + BLUP 조합이 가장 흔하게 사용됨.
목적함수를 유도하고 이를 미분하여 함수값의 최소조건으로 혼합모델방정식(MME, Mixed Model Equation)을 도출
목적함수 $Q(\boldsymbol{\beta}, \,\mathbf{a})$의 유도 과정
1. 선형혼합모델
$$\mathbf{y} = \mathbf{X} \boldsymbol{\beta} + \mathbf{Z}_a \mathbf{a} + \boldsymbol{\varepsilon}$$
여기서, $\mathbf{a} \sim \mathcal{N}(0, \mathbf{G}_a)$
$\boldsymbol{\varepsilon} \sim \mathcal{N}(0, \mathbf{R})$
2. $\mathbf{y}$의 조건부분포
$$\mathbf{y} \mid \mathbf{a} \sim \mathcal{N}(\mathbf{X} \boldsymbol{\beta} + \mathbf{Z}_a \mathbf{a}, \mathbf{R})$$
3. $\mathbf{a}$의 사전분포
$$ \mathbf{a} \sim \mathcal{N}(0, \mathbf{G}_a)$$
4. 결합우도(joint likelihood)
$$f(\mathbf{y}, \mathbf{a} \mid \boldsymbol{\beta}) = f(\mathbf{y} \mid \mathbf{a}, \boldsymbol{\beta}) \cdot f(\mathbf{a})$$
5. 결합우도에 로그를 취하면
$$\log L = -\frac{1}{2} (\mathbf{y} – \mathbf{X} \boldsymbol{\beta} – \mathbf{Z}_a \mathbf{g})^{\top} \mathbf{R}^{-1} (\mathbf{y} – \mathbf{X} \boldsymbol{\beta} – \mathbf{Z}_a \mathbf{a})
– \frac{1}{2} \mathbf{a}^{\top} \mathbf{G}_a^{-1} \mathbf{a} + \text{const}$$
6. 목적함수 $Q(\boldsymbol{\beta}, \,\mathbf{a})$로 정리
$$Q(\boldsymbol{\beta}, \,\mathbf{a}) = (\mathbf{y} – \mathbf{X} \boldsymbol{\beta} – \mathbf{Z_aa})^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{X} \beta – \mathbf{Z_aa}) + \mathbf{a}^\top \mathbf{G_a}^{-1} \mathbf{a}$$
목적함수 $Q(\boldsymbol{\beta}, \, \mathbf{a})$의 앞쪽 항은 오차항의 가중제곱합, 뒤쪽 항은 확률효과의 정규분포 페널티. 이 목적함수를 미분하여 MME를 유도. MME(혼합모델방정식, Mixed Model Equation)는 고정효과와 확률효과를 동시에 추정하기 위한 시스템. 선형 모델과 정규분포 가정, 선형최소제곱 또는 우도최적화로부터 유도됨
목적함수(Q)를 편미분하여 0으로 설정
$\boldsymbol{\beta}$로 편미분
$$-2 \mathbf{X}^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{X}\boldsymbol{\beta}- \mathbf{Z_aa}) = 0 \,\, \Rightarrow$$
$$\mathbf{X}^\top \mathbf{R}^{-1} \mathbf{X} \beta + \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{Z_aa}
= \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{y}$$
$\mathbf{a}$로 편미분
$$-2 \mathbf{Z}^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{X \beta} – \mathbf{Z_aa})
+ 2 \mathbf{G_a^{-1} a} = 0 \,\, \Rightarrow$$
$$\mathbf{Z_a}^\top \mathbf{R}^{-1} \mathbf{X \beta}
+ (\mathbf{Z_a}^\top \mathbf{R}^{-1} \mathbf{Z_a} + \mathbf{A}^{-1})\mathbf{a}
= \mathbf{Z_a}^\top \mathbf{R}^{-1} \mathbf{y}$$
편미분한 두 식을 행렬로 정리 -> MME 도출
MME(혼합모델방정식, Mixed Model Equation)는 다음식으로 표현됨
$$
\begin{bmatrix}
\mathbf{X}^\top \mathbf{R}^{-1} \mathbf{X} & \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{Z_a} \\
\mathbf{Z_a}^\top \mathbf{R}^{-1} \mathbf{X} & \mathbf{Z_a}^\top \mathbf{R}^{-1} \mathbf{Z_a} + \mathbf{G_a}^{-1}
\end{bmatrix}
\begin{bmatrix}
\hat{\boldsymbol{\beta}} \\
\hat{\mathbf{a}}
\end{bmatrix}
=
\begin{bmatrix}
\mathbf{X}^\top \mathbf{R}^{-1} \mathbf{y} \\
\mathbf{Z_a}^\top \mathbf{R}^{-1} \mathbf{y}
\end{bmatrix}
$$
여기서, $\mathbf{G_a}$는 $\sigma_a^2 \mathbf{A}$
고정효과 방정식
$$\mathbf{X}^\top \mathbf{R}^{-1} \mathbf{X} \hat{\boldsymbol{\beta}} + \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{Z_a} \hat{\mathbf{a}} = \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{y}$$
고정효과 방정식에서 $\hat{\boldsymbol{\beta}}$로 정리
$$\hat{\boldsymbol{\beta}} = \left( \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{X} \right)^{-1} \mathbf{X}^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{Z_a} \hat{\mathbf{a}})$$
윗식은 고정효과 $\boldsymbol{\beta}$의 추정량 $\hat{\boldsymbol{\beta}}$을 확률효과 $\hat{\mathbf{a}}$를 알고 있을 때의 조건부 형태로 나타낸 것
확률효과 방정식
$$\mathbf{Z_a}^\top \mathbf{R}^{-1} \mathbf{X} \hat{\boldsymbol{\beta}} + \left( \mathbf{Z_a}^\top \mathbf{R}^{-1} \mathbf{Z_a} + \mathbf{G_a}^{-1} \right) \hat{\mathbf{a}} = \mathbf{Z_a}^\top \mathbf{R}^{-1} \mathbf{y}$$
$\mathbf{\hat{a}}$식에 $\mathbf{\hat{\beta}}$가 포함된 형태로 육종가를 정리
$$\left( \mathbf{Z_a}^\top \mathbf{R}^{-1} \mathbf{Z_a} + \mathbf{G_a}^{-1} \right) \hat{\mathbf{a}} = \mathbf{Z_a}^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{X} \hat{\boldsymbol{\beta}})$$
양변에 $\left( \mathbf{Z_a}^\top \mathbf{R}^{-1} \mathbf{Z_a} + \mathbf{G_a}^{-1} \right)^{-1}$를 곱하면$$\hat{\mathbf{a}} = \left( \mathbf{Z_a}^\top \mathbf{R}^{-1} \mathbf{Z_a} + \mathbf{G_a}^{-1} \right)^{-1} \mathbf{Z_a}^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{X} \hat{\boldsymbol{\beta}})$$
육종가 $\hat{\mathbf{a}}$의 추정량은 BLUP이며 혼합모형방정식(MME)의 두 번째 행을 역행렬로 정리한 결과. MME에서 위의 두식처럼 불록행렬을 만들고 이를 이용하여 두 추정량을 계산하는 데 이 두 식은 서로 의존하므로 반복계산 또는 동시 해법을 사용. 또는 MME에서 역행렬을 이용해서 두 추정량을 계산하는 데 선형시스템해법(예, LU분해, Cholesky분해 등)을 사용함. 실전에서는 R, Python 등 에서 내부적으로 MME를 구성해 수치적으로 해를 구함
| 알고리즘 | 수렴 기준 | 일반 값 |
|---|---|---|
| EM-REML | 절대 변화량 or 상대 변화량 | $10^{-6}$ |
| AI-REML | 로그우도 변화 or 분산성분 상대오차 | $10^{-8}$ |
| 소프트웨어 | asreml, sommer, lme4, blupf90 등 |
기본값 또는 설정 가능 |
| 평가 방법 | 내용 |
|---|---|
| 상관계수 | $\mathrm{cor}(\mathbf{\hat{a}, y}) / \sqrt{h^2}$ |
| MSE | $\frac{1}{n} \sum (y_i – \hat{y}_i)^2$ |
| LRT | $\sigma_a^2$ 유의성 검정 |
| AIC/BIC | 모형 비교용 |
| Cross-validation | 외부 데이터로 검증 정확도 평가 |
$\mathbf{y}$: 반응변량의 관측값 벡터 (근내지방도)
$\mathbf{X}$: 고정효과(출생년도, 도체중) 설계행렬
$\mathbf{Z_a}$: 혈통 확률효과 설계행렬
$\mathbf{A}$: 혈통기반 유전관계행렬
$\mathbf{A}$행렬 구하기:
혈통 기반 유전관계행렬인 $\mathbf{A}$는 개체 간 유전적 관련성을 나타내는 행렬. 이를 계산하기 위해서는 부모 정보(pedigree)가 필요
Henderson (1975), 유전육종학자
혈통 기반 유전효과 예측
잠재적유전능력(육종가)에 기반한 육종
가축·작물 육종, 개량
계층형 조직 분석, 인사 평가, 기업가치 요소 추정
Genomic Best Linear Unbiased Prediction
벡터 모델식
$$\mathbf{y} = \mathbf{X}\boldsymbol{\beta} + \mathbf{Z_g}\mathbf{g} + \boldsymbol{\varepsilon}$$
$$\mathbf{y} \sim \mathcal{N}(\mathbf{X}\boldsymbol{\beta},\; \mathbf{ZG_gZ}^\top \sigma_g^2 +\mathbf{I}\sigma_e^2)$$
여기서, $\mathbf{y} \in \mathbb{R}^{n \times 1}$는 표현형(관측값) 벡터 (예: 근내지방도)
$\mathbf{X} \in \mathbb{R}^{n \times p}$는 고정효과 디자인 행렬 (예: 출생연도)
$\boldsymbol{\beta} \in \mathbb{R}^{p \times 1}$는 고정효과 계수
$\mathbf{Z_g} \in \mathbb{R}^{n \times q}$는 개체-유전효과 매핑 행렬 (보통 단위 행렬)
$\mathbf{G} \in \mathbb{R}^{q \times q}$는 유전체 유사도행렬 (Genomic relationship matrix)
$\sigma^2_g$는 유전효과의 분산 (random effect variance)
$\sigma^2_e$는 환경 오차의 분산
$\mathbf{I}$는 단위행렬 (오차 간 독립성 가정)
다변량 정규분포를 나타내는 벡터와 그 벡터의 선형변환
오차항: $\boldsymbol{\varepsilon}$
$$\boldsymbol{\varepsilon}$$
$$\boldsymbol{\varepsilon} \sim \mathcal{N}(0, \sigma_{\varepsilon}^2\mathbf{I})$$
랜덤덤효과 벡터: $\mathbf{g}$
$$\mathbf{g}$$
$$\mathbf{g} \sim \mathcal{N}(0, \sigma_g^2\mathbf{G})$$
반응변량: $\mathbf{y}$
$$\mathbf{y} = \mathbf{X}\boldsymbol{\beta} + \mathbf{Z_g}\mathbf{g} + \boldsymbol{\varepsilon}$$
$$\mathbf{y} \sim \mathcal{N}(\mathbf{X}\boldsymbol{\beta},\ \sigma^2_g \mathbf{Z_gGZ_g}^\top + \sigma^2_\varepsilon \mathbf{I})$$
BLUE: $\hat{\boldsymbol{\beta}}$
$$\hat{\boldsymbol{\beta}} = (\mathbf{X}^\top \mathbf{X})^{-1} \mathbf{X}^\top \mathbf{y}$$
$$\hat{\boldsymbol{\beta}} \sim \mathcal{N} \left( \boldsymbol{\beta},\ \sigma^2 (\mathbf{X}^\top \mathbf{X})^{-1} \right)$$
BLUP: $\hat{\mathbf{g}}$
$$\hat{\mathbf{g}} = \sigma^2_g \mathbf{G} \mathbf{Z_g}^\top \mathbf{V_g}^{-1} (\mathbf{y} – \mathbf{X} \hat{\boldsymbol{\beta}})$$
$$\hat{\mathbf{g}} \sim \mathcal{N} \left( \mathbf{g},\ \mathbf{C_gV_gC_g}^\top \right)$$
여기서, $\mathbf{C_g} = \sigma_g^2 \mathbf{A} \mathbf{Z_g}^\top \mathbf{V}^{-1} \left( \mathbf{I} – \mathbf{X}(\mathbf{X}^\top \mathbf{V}^{-1} \mathbf{X})^{-1} \mathbf{X}^\top \mathbf{V}^{-1} \right)$
| 용어 | 설명 | 의미 |
|---|---|---|
| Genomic-based | 유전체 기반 | 예측 시 유전자의 마커 정보를 이용하며, 개체 간 유전적 유사도는 유전체 정보를 기반으로 한 관계행렬 $\boldsymbol{G}$를 사용함 |
| Best | 최선의 (최소 분산) | 동일한 조건의 다른 예측량보다 분산이 가장 작다 |
| Linear | 선형 | 예측량 $\hat{\boldsymbol{g}}$는 관측값 $\boldsymbol{y}$의 선형결합으로 표현된다. $$ \begin{bmatrix} \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{X} & \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{Z} \\ \mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{X} & \mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{Z} + \mathbf{G}^{-1} \end{bmatrix} \begin{bmatrix} \hat{\boldsymbol{\beta}} \\ \hat{\mathbf{g}} \end{bmatrix} = \begin{bmatrix} \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{y} \\ \mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{y} \end{bmatrix} $$ |
| Unbiased | 불편 | 기대값이 진짜 육종가와 같다 $\mathbb{E}[\hat{g}] = g$ |
| Prediction | 예측량 | 유전체 정보를 바탕으로 개체의 유전적 가치를 예측한 값 |
모수와 모수를 추정하는 방법과 추정량
GBLUP은 MME를 통해 고정효과와 확률효과를 동시에 추정
분산성분은 REML 등으로 추정
| 모수 | 추정방법 | 추정량 |
|---|---|---|
회귀계수벡터(고정효과벡터) $\boldsymbol{\beta}$ | 혼합모형방정식(MME) 이용한 고정효과 추정 | $\hat{\boldsymbol{\beta}} = \left( \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{X} \right)^{-1} \mathbf{X}^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{Z_g} \hat{\mathbf{g}})$ |
유전체 육종가벡터(확률효과벡터) $\mathbf{g}$ | 혼합모형방정식(MME) 이용한 BLUP (확률효과) | $\hat{\mathbf{g}} = \left( \mathbf{Z_g}^\top \mathbf{R}^{-1} \mathbf{Z_g} + \mathbf{G_g}^{-1} \right)^{-1} \mathbf{Z_g}^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{X} \hat{\boldsymbol{\beta}})$ 여기서, $\mathbf{G_g} = \sigma_g^2 \mathbf{G}$ |
오차항 분산성분 $\sigma_{\varepsilon}^2$ | REML 또는 잔차제곱합 기반 추정 | $\hat{\sigma}_e^2 = \dfrac{\mathbf{e}^\top \mathbf{e}}{n – p}$ 여기서, $n$은 관측값 개수 $p$는 고정효과의 수 |
유전체기반 육종가 분산성분 $\sigma_a^2$ | REML로 분산성분 추정 | 추정량은 없고 최대우도 기반 반복 알고리즘 사용 (예: EM-REML) |
모델
$$\mathbf{y} = \mathbf{X} \boldsymbol{\beta} + \mathbf{Z_g} \mathbf{g} + \boldsymbol{\varepsilon}$$
여기서, $\mathbf{y} \sim N(\mathbf{X} \boldsymbol{\beta}, \mathbf{V_g})$
$\mathbf{V_g}$는 $\mathbf{y}$의 공분산행렬: $\mathbf{V_g}=\mathbf{Z_g} \mathbf{G_g} \mathbf{Z_g}^\top + \mathbf{R}$
$\mathbf{G_g}$는 유전체육종가의 공분산행렬: $\mathbf{G_g}=\sigma_g^2\mathbf{G}$
$\mathbf{R}$은 오차항(잔차)의 공분산행렬: $\mathbf{R} = \sigma_\varepsilon^2 \mathbf{I}$
GBLUP는 최소제곱법 만으로는 적절히 수행할 수 없으며, 혼합모형 해법(BLUP)과 분산 추정을 위한 MLE/REML이 모두 필요. 특히 REML + BLUP 조합이 가장 흔하게 사용됨.
목적함수를 유도하고 이를 미분하여 함수값의 최소조건으로 혼합모델방정식(MME, Mixed Model Equation)을 도출
목적함수 $Q(\boldsymbol{\beta}, \,\mathbf{g})$의 유도 과정
1. 선형혼합모델
$$\mathbf{y} = \mathbf{X} \boldsymbol{\beta} + \mathbf{Z}_g \mathbf{g} + \boldsymbol{\varepsilon}$$
여기서, $\mathbf{g} \sim \mathcal{N}(0, \mathbf{G}_g)$
$\boldsymbol{\varepsilon} \sim \mathcal{N}(0, \mathbf{R})$
2. $\mathbf{y}$의 조건부분포
$$\mathbf{y} \mid \mathbf{g} \sim \mathcal{N}(\mathbf{X} \boldsymbol{\beta} + \mathbf{Z}_g \mathbf{g}, \mathbf{R})$$
3. $\mathbf{g}$의 사전분포
$$ \mathbf{g} \sim \mathcal{N}(0, \mathbf{G}_g)$$
4. 결합우도(joint likelihood)
$$f(\mathbf{y}, \mathbf{g} \mid \boldsymbol{\beta}) = f(\mathbf{y} \mid \mathbf{g}, \boldsymbol{\beta}) \cdot f(\mathbf{g})$$
5. 결합우도에 로그를 취하면
$$\log L = -\frac{1}{2} (\mathbf{y} – \mathbf{X} \boldsymbol{\beta} – \mathbf{Z}_g \mathbf{g})^{\top} \mathbf{R}^{-1} (\mathbf{y} – \mathbf{X} \boldsymbol{\beta} – \mathbf{Z}_g \mathbf{g})
– \frac{1}{2} \mathbf{g}^{\top} \mathbf{G}_g^{-1} \mathbf{g} + \text{const}$$
6. 목적함수 $Q(\boldsymbol{\beta}, \,\mathbf{g})$로 정리
$$Q(\boldsymbol{\beta}, \,\mathbf{g}) = (\mathbf{y} – \mathbf{X} \boldsymbol{\beta} – \mathbf{Z_gg})^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{X} \beta – \mathbf{Z_gg}) + \mathbf{g}^\top \mathbf{G_g}^{-1} \mathbf{g}$$
목적함수 $Q(\boldsymbol{\beta}, \, \mathbf{g})$의 앞쪽 항은 오차항의 가중제곱합, 뒤쪽 항은 확률효과의 정규분포 페널티. 이 목적함수를 미분하여 MME를 유도. MME(혼합모델방정식, Mixed Model Equation)는 고정효과와 확률효과를 동시에 추정하기 위한 시스템. 선형 모델과 정규분포 가정, 선형최소제곱 또는 우도최적화로부터 유도됨
목적함수(Q)를 편미분하여 0으로 설정
$\boldsymbol{\beta}$로 편미분
$$-2 \mathbf{X}^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{X}\boldsymbol{\beta}- \mathbf{Z_gg}) = 0 \,\, \Rightarrow$$
$$\mathbf{X}^\top \mathbf{R}^{-1} \mathbf{X} \beta + \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{Z_gg}
= \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{y}$$
$\mathbf{g}$로 편미분
$$-2 \mathbf{Z}^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{X \beta} – \mathbf{Z_gg})
+ 2 \mathbf{G^{-1} g} = 0 \,\, \Rightarrow$$
$$\mathbf{Z_g}^\top \mathbf{R}^{-1} \mathbf{X \beta}
+ (\mathbf{Z_g}^\top \mathbf{R}^{-1} \mathbf{Z_g} + \mathbf{G}^{-1})\mathbf{g}
= \mathbf{Z_g}^\top \mathbf{R}^{-1} \mathbf{y}$$
편미분한 두 식을 행렬로 정리 -> MME 도출
MME(혼합모델방정식, Mixed Model Equation)는 다음식으로 표현됨
$$
\begin{bmatrix}
\mathbf{X}^\top \mathbf{R}^{-1} \mathbf{X} & \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{Z_g} \\
\mathbf{Z_g}^\top \mathbf{R}^{-1} \mathbf{X} & \mathbf{Z_g}^\top \mathbf{R}^{-1} \mathbf{Z_g} + \mathbf{G_g}^{-1}
\end{bmatrix}
\begin{bmatrix}
\hat{\boldsymbol{\beta}} \\
\hat{\mathbf{g}}
\end{bmatrix}
=
\begin{bmatrix}
\mathbf{X}^\top \mathbf{R}^{-1} \mathbf{y} \\
\mathbf{Z_g}^\top \mathbf{R}^{-1} \mathbf{y}
\end{bmatrix}
$$
여기서, $\mathbf{G_g}$는 $\sigma_g^2 \mathbf{G}$
고정효과 방정식
$$\mathbf{X}^\top \mathbf{R}^{-1} \mathbf{X} \hat{\boldsymbol{\beta}} + \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{Z_g} \hat{\mathbf{g}} = \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{y}$$
고정효과 방정식에서 $\boldsymbol{\hat{\beta}}$로 정리
$$\hat{\boldsymbol{\beta}} = \left( \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{X} \right)^{-1} \mathbf{X}^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{Z_g} \hat{\mathbf{g}})$$
윗식은 고정효과 $\boldsymbol{\beta}$의 추정량을 확률효과 $\mathbf{\hat{g}}$를 조건으로 하는 조건부 형태로 나타낸 것
확률효과 방정식
$$\mathbf{Z_g}^\top \mathbf{R}^{-1} \mathbf{X} \hat{\boldsymbol{\beta}} + \left( \mathbf{Z_g}^\top \mathbf{R}^{-1} \mathbf{Z_g} + \mathbf{G_g}^{-1} \right) \hat{\mathbf{g}} = \mathbf{Z_g}^\top \mathbf{R}^{-1} \mathbf{y}$$
$\mathbf{\hat{g}}$식에 $\mathbf{\hat{\beta}}$가 포함된 형태로 육종가를 정리
$$\left( \mathbf{Z_g}^\top \mathbf{R}^{-1} \mathbf{Z_g} + \mathbf{G_g}^{-1} \right) \hat{\mathbf{g}} = \mathbf{Z_g}^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{X} \hat{\boldsymbol{\beta}})$$
양변에 $\left( \mathbf{Z_g}^\top \mathbf{R}^{-1} \mathbf{Z_g} + \mathbf{G_g}^{-1} \right)^{-1}$를 곱하면$$\hat{\mathbf{g}} = \left( \mathbf{Z_g}^\top \mathbf{R}^{-1} \mathbf{Z_g} + \mathbf{G_g}^{-1} \right)^{-1} \mathbf{Z_g}^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{X} \hat{\boldsymbol{\beta}})$$
육종가 $\hat{\mathbf{g}}$의 추정량은 BLUP이며 혼합모형방정식(MME)의 두 번째 행을 역행렬로 정리한 결과. MME에서 위의 두식처럼 불록행렬을 만들고 이를 이용하여 두 추정량을 계산하는 데 이 두 식은 서로 의존하므로 반복계산 또는 동시 해법을 사용. 또는 MME에서 역행렬을 이용해서 두 추정량을 계산하는 데 선형시스템해법(예, LU분해, Cholesky분해 등)을 사용함. 실전에서는 R, Python 등 에서 내부적으로 MME를 구성해 수치적으로 해를 구함
REML (제한최대우도, Restricted Maximum Likelihood) 추정 과정
PBLUP에서의 분산행렬($\mathbf{G_g}$, $\mathbf{R}$)과 분산성분($\sigma_g^2$, $\sigma_e^2$)
$$\mathbf{G_g} = \sigma_g^2 \mathbf{G}, \quad \mathbf{R} = \sigma_e^2 \mathbf{I}$$
분산성분, 회귀계수벡터, 육종가벡터를 REML으로 추정하는 과정
분산성분 수렴기준(convergence criterion)
| 알고리즘 | 수렴 기준 | 일반 값 |
|---|---|---|
| EM-REML | 절대 변화량 or 상대 변화량 | $10^{-6}$ |
| AI-REML | 로그우도 변화 or 분산성분 상대오차 | $10^{-8}$ |
| 소프트웨어 | asreml, sommer, lme4, blupf90 등 | 기본값 또는 설정 가능 |
| 평가 방법 | 내용 |
|---|---|
| 상관계수 | $\mathrm{cor}(\mathbf{\hat{g}, y}) / \sqrt{h^2}$ |
| MSE | $\frac{1}{n} \sum (y_i – \hat{y}_i)^2$ |
| LRT | $\sigma_g^2$ 유의성 검정 |
| AIC/BIC | 모형 비교용 |
| Cross-validation | 외부 데이터로 검증 정확도 평가 |
$\mathbf{y}$: 반응변량 관측값 벡터 (근내지방도)
$\mathbf{X}$: 고정효과(출생년도, 도체중) 설계행렬
$\mathbf{Z_g}$: 유전체 확률효과 설계행렬
$\mathbf{G}$: 유전체기반 유전관계행렬
G행렬 구하기: VanRaden Method
$$\mathbf{G} = \frac{ZZ^\top}{2 \sum p_j (1 – p_j)}$$
여기서, $p_j$는 $j$번째 SNP에 대한 대립유전자 빈도 (minor allele frequency)
G행렬 계산
1. SNP genotype matrix $\mathbf{M}$ 입력
$$\mathbf{M}$$
여기서, $\mathbf{M}$은 $n\times m$ 행렬
$n$은 개체수
$m$은 SNP개수
원소 0은 homozygous reference AA
원소 1은 heterozygous AB
원소 2는 homozygous alternate BB
2. 중심화된 유전형 행렬 $\mathbf{Z}$ 계산
$$Z_{ij} = M_{ij} – 2p_j$$
3. 정규화 상수 c 계산
$$c = 2 \sum_{j=1}^{m} p_j(1 – p_j)$$
4. 유전체행렬 $\mathbf{G}$ 계산
$$\mathbf{G} = \frac{ZZ^\top}{c}$$
5. 위의 1. ~ 4.를 결합하여 한 줄로 표현
$$\mathbf{G} = \frac{(M – 2p)(M – 2p)^\top}{2 \sum\limits_{j=1}^{m} p_j(1 – p_j)}$$
VanRaden (2008), 유전육종학자
유전체 기반 유전효과 예측
잠재적유전능력(육종가)에 기반한 육종
유전체육종, 유전평가
고객 행동 기반 주가 반응 예측, 마케팅 ROI 분석
Single-Step Genomic Best Linear Unbiased Prediction
벡터 모델식
$$\mathbf{y} = \mathbf{X}\boldsymbol{\beta} + \mathbf{Z}\mathbf{u} + \boldsymbol{\varepsilon} \quad , \quad \mathbf{y} \sim \mathcal{N}(\mathbf{X}\boldsymbol{\beta},\; \mathbf{ZHZ}^\top \sigma_u^2 + I\sigma_{\varepsilon}^2)$$
여기서, $\mathbf{y} \in \mathbb{R}^{n \times 1}$는 표현형(관측값) 벡터 (예: 근내지방도, 도체중 등)
$\mathbf{X} \in \mathbb{R}^{n \times p}$는 고정효과 디자인 행렬 (예: 성별, 연도, 지역 등)
$\boldsymbol{\beta} \in \mathbb{R}^{p \times 1}$는 고정효과 계수
$\mathbf{Z} \in \mathbb{R}^{n \times q}$는 개체별 유전효과를 연결하는 매핑 행렬
$\mathbf{H} \in \mathbb{R}^{q \times q}$는 혈통관계행렬 $\mathbf{A}$과 유전체관계행렬
$\mathbf{G}$을 통합한 유전관계행렬
$\sigma^2_u$는 유전효과의 분산 (random effect variance)
$\sigma_{\varepsilon}^2$는 오차항 분산 (환경효과 포함)
$\mathbf{I}$는 단위행렬 (오차 간 독립성 가정)
$$\mathbf{H}^{-1} = A^{-1} +
\begin{bmatrix}
0 & 0 \\
0 & \mathbf{G}^{-1} – \mathbf{A}_{22}^{-1}
\end{bmatrix}$$
여기서, $\mathbf{A}^{-1}$는 전체 개체의 혈통기반 관계 역행렬
$G^{-1}$는 유전체 정보가 있는 개체의 유전체 관계 역행렬
$\mathbf{A}_{22}^{-1}$는 그 유전체 개체들의 혈통기반 하위행렬 역행렬
$\mathbf{H}$의 실제 사용 시, $\mathbf{H}^{-1}$을 구성하여 REML 등에서 사용
$\mathbf{H} \in \mathbb{R}^{n \times n}$
$n$은 전체 개체수
다변량 정규분포를 나타내는 벡터와 그 벡터의 선형변환
오차항: $\boldsymbol{\varepsilon}$
$$\boldsymbol{\varepsilon}$$
$$\boldsymbol{\varepsilon} \sim \mathcal{N}(0, \sigma_{\varepsilon}^2\mathbf{I})$$
확률효과 벡터: $\mathbf{u}$
$$\mathbf{u}$$
$$\mathbf{u} \sim \mathcal{N}(0, \sigma_u^2\mathbf{H})$$
반응변량: $\mathbf{y}$
$$\mathbf{y} = \mathbf{X}\boldsymbol{\beta} + \mathbf{Z}\mathbf{u} + \boldsymbol{\varepsilon}$$
$$\mathbf{y} \sim \mathcal{N}(\mathbf{X}\boldsymbol{\beta},\ \sigma^2_u \mathbf{ZHZ}^\top + \sigma^2_\varepsilon \mathbf{I})$$
BLUE: $\hat{\boldsymbol{\beta}}$
$$\hat{\boldsymbol{\beta}} = (\mathbf{X}^\top \mathbf{X})^{-1} \mathbf{X}^\top \mathbf{y}$$
$$\hat{\boldsymbol{\beta}} \sim \mathcal{N} \left( \boldsymbol{\beta},\ \sigma^2 (\mathbf{X}^\top \mathbf{X})^{-1} \right)$$
BLUP: $\hat{\mathbf{u}}$
$$\hat{\mathbf{u}} = \sigma^2_u \mathbf{H} \mathbf{Z}^\top \mathbf{V}^{-1} (\mathbf{y} – \mathbf{X} \hat{\boldsymbol{\beta}})$$
$$\hat{\mathbf{u}} \sim \mathcal{N} \left( \mathbf{u},\ \mathbf{CVC}^\top \right)$$
여기서, $\mathbf{C} = \sigma_h^2 \mathbf{H} \mathbf{Z}^\top \mathbf{V}^{-1} \left( \mathbf{I} – \mathbf{X}(\mathbf{X}^\top \mathbf{V}^{-1} \mathbf{X})^{-1} \mathbf{X}^\top \mathbf{V}^{-1} \right)$
ssBLUP은 MME를 통해 고정효과와 확률효과를 동시에 추정
분산성분은 REML 등으로 추정
| 모수 | 추정방법 | 추정량 |
|---|---|---|
| 회귀계수벡터 (고정효과벡터) $\boldsymbol{\beta}$ | 혼합모형방정식(MME) 이용한 고정효과 추정 | $\hat{\boldsymbol{\beta}} = \left( \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{X} \right)^{-1} \mathbf{X}^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{Z} \hat{\mathbf{u}})$ |
| 육종가벡터 (확률효과벡터) $\mathbf{u}$ | 혼합모형방정식(MME) 이용한 확률효과 추정 | $\hat{\mathbf{u}} = \left( \mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{Z} + \mathbf{G}^{-1} \right)^{-1} \mathbf{Z}^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{X} \hat{\boldsymbol{\beta}})$
여기서, $\mathbf{G} = \sigma_u^2 \mathbf{H}$ |
| 오차항 분산성분 $\sigma_{\varepsilon}^2$ | REML 또는 잔차제곱합 기반 추정 | $\hat{\sigma}_e^2 = \dfrac{\mathbf{e}^\top \mathbf{e}}{n – p}$
여기서, $n$은 관측값 개수 $p$는 고정효과의 수 |
| 육종가 분산성분 $\sigma_u^2$ | REML로 분산성분 추정 | 추정량은 없고 최대우도 기반 반복 알고리즘 사용 (예: EM-REML) |
모델
$$\mathbf{y} = \mathbf{X} \boldsymbol{\beta} + \mathbf{Z} \mathbf{u} + \boldsymbol{\varepsilon}$$
여기서, $\mathbf{y} \sim N(\mathbf{X} \boldsymbol{\beta}, \mathbf{V})$
$\mathbf{V}$는 $\mathbf{y}$의 공분산행렬: $\mathbf{V}=\mathbf{Z} \mathbf{G} \mathbf{Z}^\top + \mathbf{R}$
$\mathbf{G}$는 유전체육종가의 공분산행렬: $\mathbf{G}=\sigma_g^2\mathbf{G}$
$\mathbf{R}$은 오차항(잔차)의 공분산행렬: $\mathbf{R} = \sigma_\varepsilon^2 \mathbf{I}$
GBLUP는 최소제곱법 만으로는 적절히 수행할 수 없으며, 혼합모형 해법(BLUP)과 분산 추정을 위한 MLE/REML이 모두 필요. 특히 REML + BLUP 조합이 가장 흔하게 사용됨.
목적함수를 유도하고 이를 미분하여 함수값의 최소조건으로 혼합모델방정식(MME, Mixed Model Equation)을 도출
목적함수 $Q(\boldsymbol{\beta}, \,\mathbf{g})$의 유도 과정
1. 선형혼합모델
$$\mathbf{y} = \mathbf{X} \boldsymbol{\beta} + \mathbf{Z} \mathbf{u} + \boldsymbol{\varepsilon}$$
여기서, $\mathbf{u} \sim \mathcal{N}(0, \mathbf{G})$
$\boldsymbol{\varepsilon} \sim \mathcal{N}(0, \mathbf{R})$
2. $\mathbf{y}$의 조건부분포
$$\mathbf{y} \mid \mathbf{u} \sim \mathcal{N}(\mathbf{X} \boldsymbol{\beta} + \mathbf{Z} \mathbf{g}, \mathbf{R})$$
3. $\mathbf{g}$의 사전분포
$$ \mathbf{g} \sim \mathcal{N}(0, \mathbf{G})$$
4. 결합우도(joint likelihood)
$$f(\mathbf{y}, \mathbf{u} \mid \boldsymbol{\beta}) = f(\mathbf{y} \mid \mathbf{u}, \boldsymbol{\beta}) \cdot f(\mathbf{u})$$
5. 결합우도에 로그를 취하면
$$\log L = -\frac{1}{2} (\mathbf{y} – \mathbf{X} \boldsymbol{\beta} – \mathbf{Z} \mathbf{g})^{\top} \mathbf{R}^{-1} (\mathbf{y} – \mathbf{X} \boldsymbol{\beta} – \mathbf{Z} \mathbf{u})
– \frac{1}{2} \mathbf{u}^{\top} \mathbf{G}^{-1} \mathbf{u} + \text{const}$$
6. 목적함수 $Q(\boldsymbol{\beta}, \,\mathbf{u})$로 정리
$$Q(\boldsymbol{\beta}, \,\mathbf{u}) = (\mathbf{y} – \mathbf{X} \boldsymbol{\beta} – \mathbf{Zu})^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{X} \beta – \mathbf{Zu}) + \mathbf{u}^\top \mathbf{G}^{-1} \mathbf{u}$$
목적함수 $Q(\boldsymbol{\beta}, \, \mathbf{u})$의 앞쪽 항은 오차항의 가중제곱합, 뒤쪽 항은 확률효과의 정규분포 페널티. 이 목적함수를 미분하여 MME를 유도. MME(혼합모델방정식, Mixed Model Equation)는 고정효과와 확률효과를 동시에 추정하기 위한 시스템. 선형 모델과 정규분포 가정, 선형최소제곱 또는 우도최적화로부터 유도됨
목적함수(Q)를 편미분하여 0으로 설정
$\boldsymbol{\beta}$로 편미분
$$-2 \mathbf{X}^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{X}\boldsymbol{\beta}- \mathbf{Zu}) = 0 \,\, \Rightarrow$$
$$\mathbf{X}^\top \mathbf{R}^{-1} \mathbf{X} \beta + \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{Zu}
= \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{y}$$
$\mathbf{g}$로 편미분
$$-2 \mathbf{Z}^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{X \beta} – \mathbf{Zu})
+ 2 \mathbf{G^{-1} g} = 0 \,\, \Rightarrow$$
$$\mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{X \beta}
+ (\mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{Z} + \mathbf{A}^{-1})\mathbf{u}
= \mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{y}$$
편미분한 두 식을 행렬로 정리 -> MME 도출
MME(혼합모델방정식, Mixed Model Equation)는 다음식으로 표현됨
$$
\begin{bmatrix}
\mathbf{X}^\top \mathbf{R}^{-1} \mathbf{X} & \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{Z} \\
\mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{X} & \mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{Z} + \mathbf{H}^{-1}
\end{bmatrix}
\begin{bmatrix}
\hat{\boldsymbol{\beta}} \\
\hat{\mathbf{u}}
\end{bmatrix}
=
\begin{bmatrix}
\mathbf{X}^\top \mathbf{R}^{-1} \mathbf{y} \\
\mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{y}
\end{bmatrix}
$$
여기서, $\mathbf{H}$는 $\sigma_u^2 \mathbf{H}$
고정효과 방정식
$$\mathbf{X}^\top \mathbf{R}^{-1} \mathbf{X} \hat{\boldsymbol{\beta}} + \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{Z} \hat{\mathbf{u}} = \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{y}$$
고정효과 방정식에서 $\mathbf{\hat{\beta}}$로 정리
$$\hat{\boldsymbol{\beta}} = \left( \mathbf{X}^\top \mathbf{R}^{-1} \mathbf{X} \right)^{-1} \mathbf{X}^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{Z} \hat{\mathbf{u}})$$
윗식은 고정효과 $\mathbf{\beta}$의 추정량을 확률효과 $\mathbf{\hat{u}}$를 알고 있을 때의 조건부 형태로 나타낸 것
육종가 방정식
$$\mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{X} \hat{\boldsymbol{\beta}} + \left( \mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{Z} + \mathbf{G}^{-1} \right) \hat{\mathbf{u}} = \mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{y}$$
$\mathbf{\hat{u}}$식에 $\mathbf{\hat{\beta}}$가 포함된 형태로 육종가 방정식을 정리
$$\left( \mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{Z} + \mathbf{G}^{-1} \right) \hat{\mathbf{u}} = \mathbf{Z}^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{X} \hat{\boldsymbol{\beta}})$$
양변에 $\left( \mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{Z} + \mathbf{G}^{-1} \right)^{-1}$를 곱하면$$\hat{\mathbf{u}} = \left( \mathbf{Z}^\top \mathbf{R}^{-1} \mathbf{Z} + \mathbf{G}^{-1} \right)^{-1} \mathbf{Z}^\top \mathbf{R}^{-1} (\mathbf{y} – \mathbf{X} \hat{\boldsymbol{\beta}})$$
이 수식은 육종가 $\hat{\mathbf{u}}$의 BLUP 추정식이며 혼합모형방정식(MME)의 두 번째 행을 역행렬로 정리한 결과
MME에서 위의 두식처럼 불록행렬을 만들고 이를 이용하여 두 추정량을 계산하는 데 이 두 식은 서로 의존하므로 반복계산 또는 동시 해법을 사용. 또는 MME에서 역행렬을 이용해서 두 추정량을 계산하는 데 선형시스템해법(예, LU분해, Cholesky분해 등)을 사용함. 실전에서는 R, Python 등 에서 내부적으로 MME를 구성해 수치적으로 해를 구함
REML (Restricted Maximum Likelihood) 추정 과정
ssBLUP에서의 분산행렬($\mathbf{G}$, $\mathbf{R}$)과 분산성분($\sigma_h^2$, $\sigma_e^2$)
$$\mathbf{G} = \sigma_h^2 \mathbf{H}, \quad \mathbf{R} = \sigma_e^2 \mathbf{I}$$
분산성분, 회귀계수벡터, 육종가벡터를 REML으로 추정하는 과정
분산성분 수렴기준(convergence criterion)
| 알고리즘 | 수렴 기준 | 일반 값 |
|---|---|---|
| EM-REML | 절대 변화량 or 상대 변화량 | $10^{-6}$ |
| AI-REML | 로그우도 변화 or 분산성분 상대오차 | $10^{-8}$ |
| 소프트웨어 | asreml, sommer, lme4, blupf90 등 | 기본값 또는 설정 가능 |
모델 평가
| 평가 방법 | 내용 |
|---|---|
| 상관계수 | $\mathrm{cor}(\hat{u}, y) / \sqrt{h^2}$ |
| MSE | $\frac{1}{n} \sum (y_i – \hat{y}_i)^2$ |
| LRT | $\sigma_u^2$ 유의성 검정 |
| AIC/BIC | 모형 비교용 |
| Cross-validation | 외부 데이터로 검증 정확도 평가 |
$\mathbf{y}$: 반응변량 관측값 (근내지방도)
$\mathbf{X}$: 고정효과(출생년도, 도체중) 설계행렬
$\mathbf{Z_u}$: 유전체|혈통 확률효과 설계행렬
$\mathbf{A}$= 혈통기반 유전관계행렬
$\mathbf{G}$: 유전체기반 유전관계행렬
$\mathbf{H}$: 유전체|혈통 기반 유전관계행렬
혈통기반 행렬 $\mathbf{A}$와 유전체기반 행렬 $\mathbf{G}$를 통합하여, 유전체정보가 있는 개체와 없는 개체를 동시에 모델링
$$\mathbf{H} =
\begin{bmatrix}
\mathbf{A}_{11} & \mathbf{A}_{12} \\
\mathbf{A}_{21} & \mathbf{G}
\end{bmatrix}$$
| 행렬 구분 | 설명 |
|---|---|
| $\mathbf{A}_{11}$ | 유전체 없는 개체들 간 혈통 기반 유사도 |
| $\mathbf{A}_{22}$ | 유전체 있는 개체들 간 혈통 기반 유사도 |
| $\mathbf{A}_{12}=\mathbf{A}_{21}$ | 두 그룹 간 혈통 기반 유사도 |
| $\mathbf{G}$ | 유전체 기반 유사도 |
Aguilar, Misztal et al. (2010) 유전육종학자들
혈통+유전체 통합 기반 유전효과 예측
잠재적유전능력(육종가)에 기반한 육종
국가 단위 개체평가, 실무육종
불완전 정보 보정, 부분 정보 기반 주가 산정, 복합 HR/재무 통합 분석
deeplearning Genomic Best Linear Unbiased Prediction
벡터 모델식
$$\hat{\mathbf{y}} = \bar{\mathbf{y}}_{\text{train}} + \hat{\mathbf{b}}_{\text{deep}} + \hat{\mathbf{b}}_a + \hat{\mathbf{b}}_d + \hat{\mathbf{b}}_e$$
여기서, $ \bar{\mathbf{y}}_{\text{train}}$는 훈련 데이터의 평균
$\hat{\mathbf{b}}_{\text{deep}}$ deep learning 모듈이 추정한 유전효과
$\hat{\mathbf{b}}_a$는 additive (가법) 유전효과
$\hat{\mathbf{b}}_d$는 dominance (우성) 유전효과
$\hat{\mathbf{b}}_e$는 epistasis (상호작용) 유전효과
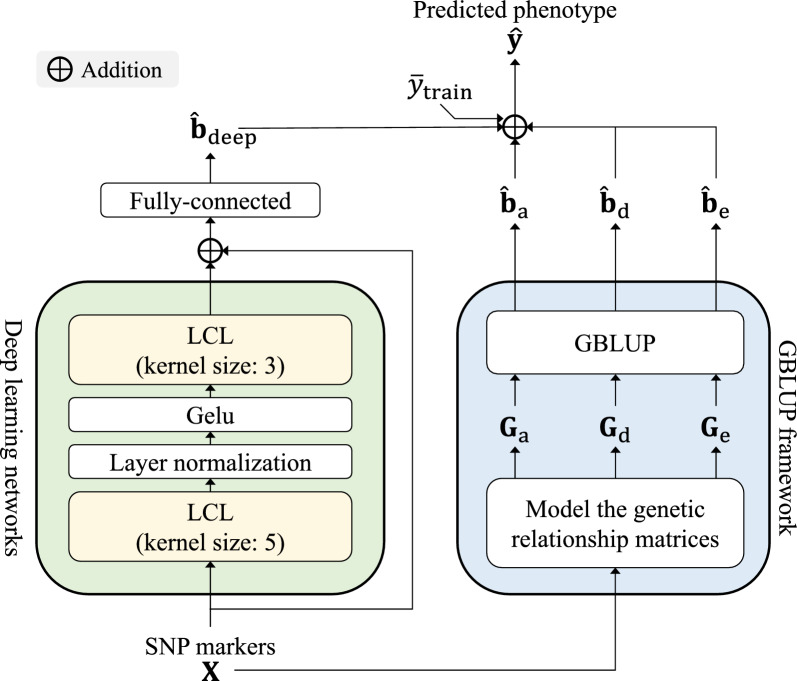
유전체 위치효과를 딥러닝으로 반영
$\hat{\mathbf{b}}_a$는 additive (가법) 유전효과
확률효과
$\hat{\mathbf{b}}_{\text{deep}}$ deep learning 모듈이 추정한 유전효과
$\hat{\mathbf{b}}_d$는 dominance (우성) 유전효과
$\hat{\mathbf{b}}_e$는 epistasis (상호작용) 유전효과
Lee SH et al. (2023) 유전육종학자들
딥러닝의 유전체의 위치에 따른 특성 추출 능력과 GBLUP의 유전행렬 기반 예측력을 통합하여 표현형 예측 성능을 향상시킨 모델.
잠재적유전능력(육종가)에 기반한 육종
LCL 기반 딥러닝 모듈이 SNP의 위치 정보를 활용
전통 GBLUP + deep feature 병렬 조합으로 예측 정확도 향상
정규분포 기반 해석과 딥러닝의 표현력을 모두 고려한 최신 예측틀
복합 HR·재무 통합 분석, 부분 정보 기반 주가 산정
유전체 기반 개체 선발, 딥러닝 기반 표현형 보완 예측